Toute sorte d’images
Plus de 315 formats, lus grâce à des bibliothèques de traitement d’image (opencv, openslide, dicom, etc.), ou transmis par des serveurs de tuiles (bioformats, google maps, etc.)
La numérisation et la visualisation d’images en anatomo-pathologie ne cesse de s’améliorer.
Cependant, aujourd’hui encore, environ 10% des images doivent être numérisées au moins deux fois et la vérification de la qualité d’une image est réalisée visuellement, par un spécialiste.
magIQ analyse automatiquement la qualité des images avec une vitesse et une précision uniques, pour une gestion plus efficace des ressources, une amélioration continue de la qualité des images et de la qualité des soins.
magIQ lit plus de 315 formats de lames virtuelles. Le logiciel navigue à travers tous les niveaux de grossissement disponibles et toutes les régions d’une image, pixel par pixel.
Il analyse ensuite les images au crible de plusieurs paramètres et calcule pour chacun de ces paramètres des scores numériques normalisés : Détection d’échantillon, mise au point, teinte, saturation, brillance, etc.
magIQ n’a pas besoin de comparer votre image à d’autres images ou à une image de référence pour en évaluer sa qualité. Il n’utilise pas vos données pour mettre à jour ses scores de qualité. Enfin, les scores de magIQ ne changent pas avec le temps.
Ces résultats sont enregistrés dans un rapport au format JSON qui regroupe l’ensemble de ces valeurs numériques et d’une cartographie de la qualité de l’image.
 Cartographie de qualité générées pour le grossissement 5x (25% de la taille originale de la lame virtuelle) en 6.67 secondes avec la configuration minimale pour un usage quotidien.
Cartographie de qualité générées pour le grossissement 5x (25% de la taille originale de la lame virtuelle) en 6.67 secondes avec la configuration minimale pour un usage quotidien. Cartographie de qualité générées pour le grossissement 10x (50% de la taille originale de la lame virtuelle) en 43.83 secondes avec la configuration minimale pour un usage quotidien.
Cartographie de qualité générées pour le grossissement 10x (50% de la taille originale de la lame virtuelle) en 43.83 secondes avec la configuration minimale pour un usage quotidien.Une fois les images analysées, magIQ les trie en trois catégories : À garder / À vérifier / À réacquérir. Les seuils pour ces trois catégories sont paramétrables : Les lames histologiques exigent généralement des seuils plus hauts que celles de lames d’immunohistochimie.
Exemple de rapport généré à partir d'un répertoire d'analyses :
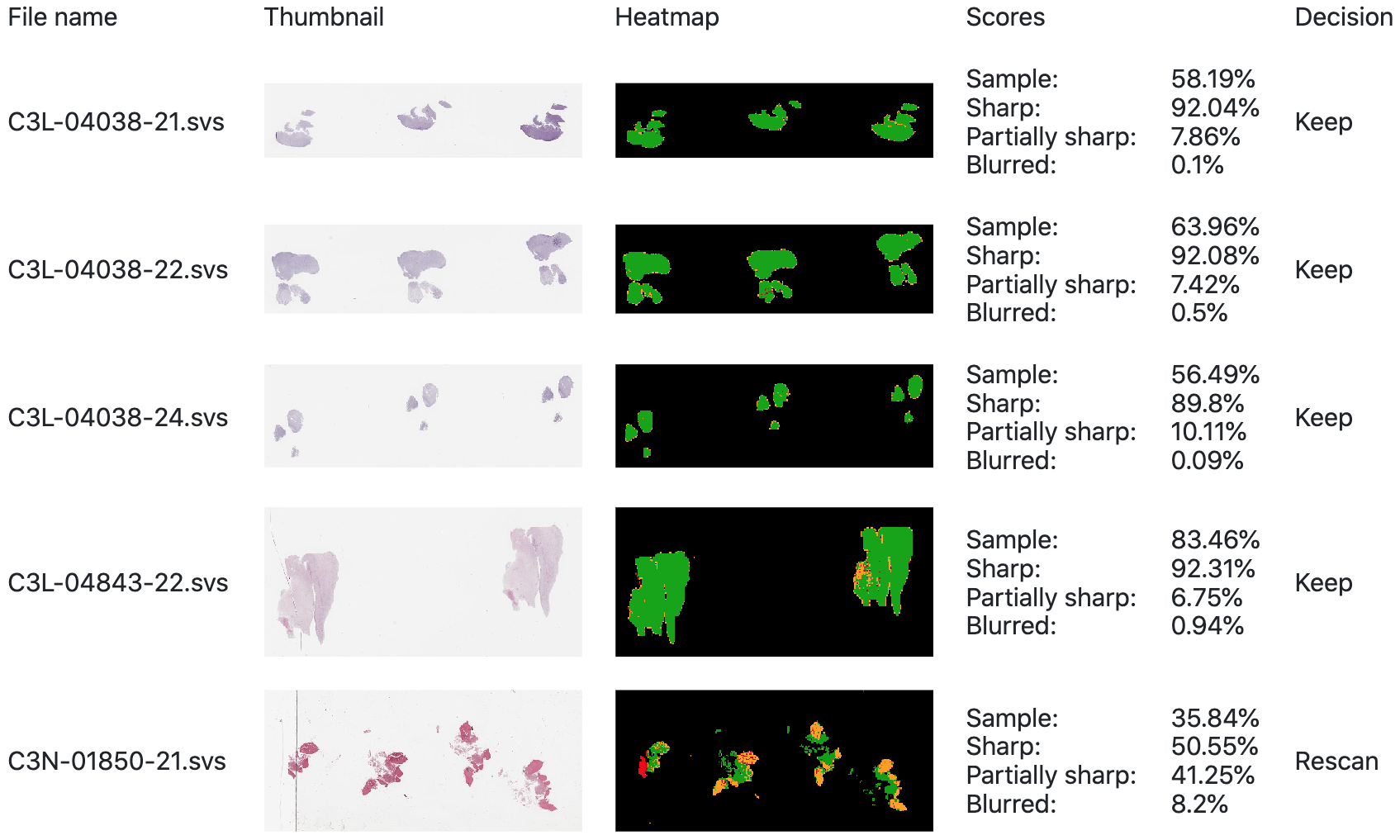
85% des images sont ainsi automatiquement acceptées ou écartées, ce qui permet de diviser par six le temps de vérification manuelle de la qualité des images.
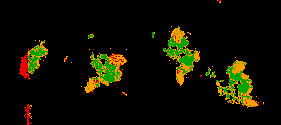
analysée au grossissement 10x
(50% de la taille originale de la lame virtuelle)
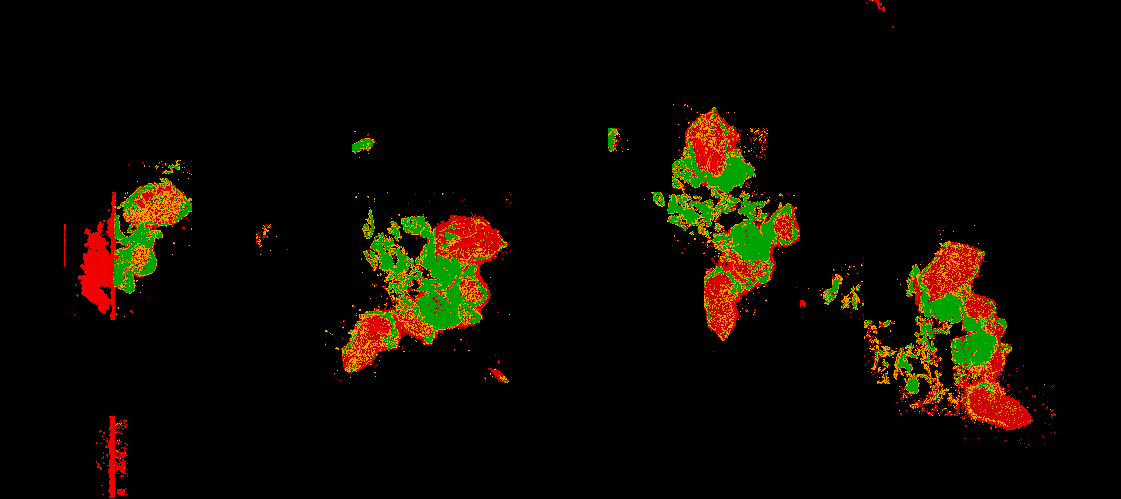
analysée au grossissement 20x
(taille originale de la lame virtuelle)
Aujourd’hui, les systèmes de pathologie numérique sont composés d’un ou plusieurs matériels d’acquisition (scanner ou microscope motorisé).
Une fois acquises, les lames virtuelles sont stockées dans un ou plusieurs serveurs et diffusées via une application de gestion et de visualisation.
Elles sont ensuite éventuellement analysées avec des outils d’analyse d’image faisant appel ou non à de l’intelligence artificielle (reconnaissance de formes, comptage de noyaux…
La qualité de l’image est vérifiée à la main, une fois les images stockées, la plupart du temps dans le contexte de l’application de gestion et de visualisation des lames virtuelles.
Par ailleurs, cette vérification n’est ni systématique ni objective : l’évaluation de la qualité varie d’un individu à l’autre, d’un service à l’autre, d’un moment à l’autre.
Une image de qualité insuffisante est donc, au mieux, détectée après stockage, au pire au moment du diagnostic par le médecin.
Cette situation est à l’origine de blocages réguliers dans le flux de travail quotidien des laboratoires. Ces blocages mobilisent les médecins, ingénieurs et techniciens, et occasionnent des dépenses considérables en termes de stockage, de temps de calcul, de bande passante et d’énergie.
Plus largement, cette absence de contrôle qualité systématique et automatisé de la qualité des images est un frein à l’usage généralisé de la pathologie numérique à des freins diagnostiques.
La numérisation a pourtant des bénéfices considérables en termes de collaboration et d’aide au diagnostic, et elle bénéficie aujourd’hui d’immenses progrès aussi bien en termes de qualité et de temps d’acquisition, qu’en terme de stockage, de diffusion, et d’analyse des images numérisées.
L’intégration de magIQ à l’appareil photo, au microscope motorisé ou au scanner de lames virtuelles permet par exemple de notifier automatiquement l’agent s’il doit ré-acquérir une image (et non 48 heures après que le spécialiste l’a reçue et ouverte).
Il permet également de notifier automatiquement et instantanément le système d’acquisition pour qu’il ré-acquière une image, en partie ou en totalité, pendant le processus d’acquisition.
Les images sont souvent hétérogènes, de différentes natures, provenant de sources différentes.
Quelle que soit leur diversité, magIQ vous aide à vérifier la qualité de l’ensemble de vos images une fois stockées pour ne conserver que celles dont vous avez besoin.
Intégré au logiciel de gestion des images de pathologie, le contrôle qualité automatique de magIQ permet un stockage, une diffusion et un transfert plus rapides et efficaces.
Intégré aux systèmes d’analyse d’image, magIQ leur permet de se concentrer uniquement sur ce qu’ils sont censés analyser pour se concentrer sur l’essentiel : le diagnostic.
Plus de 315 formats, lus grâce à des bibliothèques de traitement d’image (opencv, openslide, dicom, etc.), ou transmis par des serveurs de tuiles (bioformats, google maps, etc.)
3 milliards de pixels analysés par cœur et par minute.
Les images sont analysées plus rapidement que les suivantes ne sont créées.
Un système d’assurance qualité qui s’adapte à la taille de votre infrastructure et tire le meilleur parti de votre capacité de calcul.
Avec 10 années de recherche dans le domaine, notre R&D est aujourd’hui validée par des anatomo-pathologistes et des spécialistes d’imagerie.
Notre technologie utilise des algorithmes en instance de brevet.
Notre notation est continue et reproductible. Des images de natures différentes peuvent être triées aussi aisément que des images similaires.
La création de profils spécifiques permet d’affiner l’analyse et de conserver les seuils les plus adaptés à un même ensemble d’images.
Requiert peu de mémoire, et peu de puissance de calcul.
Fonctionne sur les configurations informatiques les plus modestes comme sur les systèmes de calcul haute performance, dans des structures de toutes tailles.
S’intègre aux flux de travail existants (l’image et son contexte en entrée, un fichier JSON de résultats et une cartographie de qualité au format PNG en sortie).
Linux, Windows, macOS.
En ligne de commande ou avec une interface graphique.
Utilisable sous la forme d’un conteneur (Docker, Singularity, etc.).
Configuration minimale du serveur :
Configuration minimale du serveur pour un usage quotidien :
Configuration recommandée du serveur pour analyser un To (1.000 Go) d’images en un jour :
Configuration optimale des serveurs pour analyser un plus grand nombre d’images encore :
Intéressés ?
Nous avons déjà analysé des millions d’images. Faites-nous parvenir quelques-unes des vôtres et nous aurons le plaisir de vous transmettre en retour un rapport d’analyse, gratuitement et en toute confidentialité.